共同通信PRワイヤー KYODO NEWS PR WIRE
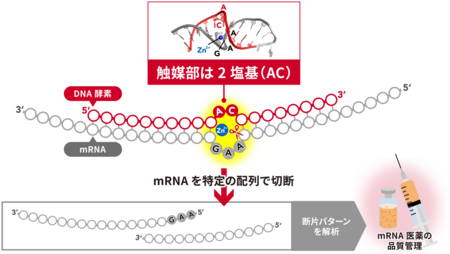
わずか2塩基でRNA切断を触媒する世界最小DNA酵素
国立研究開発法人産業技術総合研究所
ポイント
・ 2塩基の配列部分が触媒部として機能する世界最小のDNA酵素を開発
・ X線結晶構造解析とNMRスペクトル解析により、亜鉛イオンを含む反応中間体の立体構造を決定し、RNA加水分解の触媒メカニズムを解明
・ mRNAワクチンに用いられるmRNAの品質を管理するためのツールとして期待
【画像:https://kyodonewsprwire.jp/img/202601192637-O1-5WgkV0oJ】
概 要
国立研究開発法人産業技術総合研究所(以下「産総研」という)モレキュラーバイオシステム研究部門 山崎和彦 主任研究員、久保田智巳 主任研究員、健康医工学研究部門 宮岸真 キャリアリサーチャー(筑波大学グローバル教育院 教授)、猪股梨華 大学院生(筑波大学 グローバル教育院・研究当時)らの研究グループは、東京大学大学院 薬学系研究科 竹内恒 教授、近畿大学 生物理工学部 宮下尚之 准教授らと共同で、世界最小のDNA酵素を開発し、亜鉛イオンを利用したRNA加水分解の触媒メカニズムを解明しました。
DNA酵素は触媒作用を持つ人工的な一本鎖DNAで、RNAの加水分解は主な機能の一つです。標的のRNA配列に特異的に結合する基質認識部と、金属イオンの存在下で触媒作用を発揮する触媒部から構成されます。タンパク質酵素に比べて安定性が高く、切断部位の配列特異性が高いことから、RNA操作技術や医薬品開発分野での応用が期待されています。
DNA酵素は、これまでに触媒部が10〜15塩基程度のものが報告されています。今回、独自のインビトロ(in vitro)核酸選別法で開発した新規DNA酵素は、触媒部として必要な塩基数が2塩基のみであり、世界最小のDNA酵素であることが明らかになりました。また、NMRスペクトル解析とX線結晶構造解析を組み合わせた立体構造解析により、亜鉛イオンを含む反応中間体の立体構造を決定しました。これらの結果から、亜鉛イオンが配位する塩基を特定して触媒メカニズムを解明しました。
本DNA酵素は、mRNA医薬の品質管理に役立つと期待されます。数千ヌクレオチドからなるmRNAの配列を一度に決定することは困難ですが、特定の部位で切断して鎖長を解析することで、異常の有無を評価できます。今回開発したDNA酵素は、mRNA中の3塩基ほどの特定の配列を切断対象と認識するため、ほとんどのmRNAについて利用できると考えられます。
今回の研究成果の詳細は2025年1月15日に「Nucleic Acids Research」に掲載されました。
下線部は【用語解説】参照
※本プレスリリースでは、化学式や単位記号の上付き・下付き文字を、通常の文字と同じ大きさで表記しております。
正式な表記でご覧になりたい方は、産総研WEBページ
( https://www.aist.go.jp/aist_j/press_release/pr2026/pr20260121_2/pr20260121_2.html )をご覧ください。
開発の社会的背景
mRNA医薬は、感染症予防やがん治療など幅広い分野で革新的な治療法として注目されています。設計したmRNAの塩基配列に基づき、体内で目的のタンパク質を効率よく作らせることができるため、従来のワクチンや治療薬では実現が難しかった迅速な医薬品の開発や個別化医療への対応が期待されています。
一方で、mRNA医薬に利用されるような数千ヌクレオチドにおよぶ長鎖mRNAを合成・製造する過程では、設計どおりの長さのmRNAだけでなく、わずかに短いものや末端が延長されたものなど、長さの異なるmRNA分子が混在しやすいという問題があります。こうした「異なる長さのmRNA」が、どの程度含まれているかを把握し管理することが、品質管理上の大きな課題となっています。例えばmRNAワクチンにおいて、想定していない長さのmRNAが一定割合以上含まれていると、予期せぬ翻訳産物が合成されたりや副反応につながったりするリスクがあります。
現在、配列や長さの確認には次世代シーケンシングやサンガー法による配列解析が用いられていますが、長鎖mRNAを一度に詳細に読み取ることは容易ではなく、高コストかつ時間もかかるため、製造バッチごとの日常的な品質管理に用いることは現実的ではありません。
そこで、特定のRNA配列を正確に切断して解析できるDNA酵素の活用が期待されています。DNA酵素を利用することで、長鎖mRNAを狙った位置で切断し、断片パターンから「長さの異なるmRNAの混在」を簡便かつ効率的に評価できる新たなツールとして、品質管理や安全性評価への応用が可能になると考えられます。DNA酵素は、触媒として必要な非相補性配列(触媒部)と、基質であるRNAの特異的認識に関わる相補的な配列(基質認識部)からなる人工的に作られた一本鎖DNAです。酵素の基質認識部をmRNA分子の切断可能部位と適合させることにより、局所的切断ツールとして利用することができます。
研究の経緯
産総研では、これまでインビトロ核酸選別法を用いて、有用な核酸分子の開発を行ってきました(2020年10月30日産総研プレス発表)。この研究では、ゲノム編集に用いられる酵素Cas9に特異的に結合する核酸アプタマーをin vitroで選別し、その核酸アプタマーの有無によってCas9の切断活性を調節できることを示しました。これにより、ゲノム編集のスイッチを外部から入れたり切ったりできる仕組みとして、意図しない編集を抑えることが期待されています。さらに、新たなmRNA切断ツールの開発を目的として、独自開発のインビトロ核酸触媒選別法を構築し、新規DNA酵素の探索を行っています。今回発見したDNA酵素の配列情報は、既に特許第7437020号(特願 2020-49242)として発表しています。さらに本研究では、配列特異性など詳細な解析を行ってDNA酵素の特性を明らかにするとともに、触媒メカニズム解明のため、X線結晶構造解析とNMRスペクトル解析とを組み合わせた反応中間体の立体構造解析に取り組みました。
なお、本研究開発は、NEDO(国立研究開発法人新エネルギー・産業技術総合開発機構)の「植物等の生物を用いた高機能品生産技術の開発」(2016~2021年度)、文部科学省「先端研究基盤共用促進事業(先端研究設備プラットフォームプログラム)」・NMRプラットフォームJPMXS0450100021による支援を受けています。
研究の内容
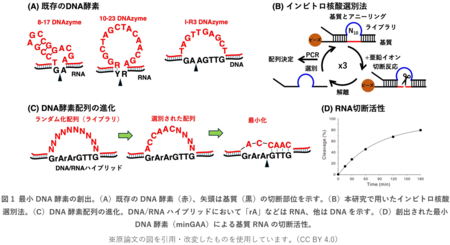
既存のDNA酵素は、10〜15塩基程度の触媒部を持ちます(図1A)。本研究では、亜鉛イオンの存在下でDNAを切断するDNA酵素I-R3を参考に、RNAを切断する新規DNA酵素の開発を行いました。酵素が基質を切断するとビーズから解離する仕組みを利用する、独自のインビトロ核酸選別法(図1B)により配列を取得し、不確定な配列部分を除去しました(図1C)。その結果、触媒部が2塩基のみという最小DNA酵素(minGAA)を取得することに成功しました。DNA/RNAハイブリッドの基質を選別に用いましたが、RNAのみからなる基質も切断できます(図1D)。
【画像:https://kyodonewsprwire.jp/img/202601192637-O2-8Yq1pT75】
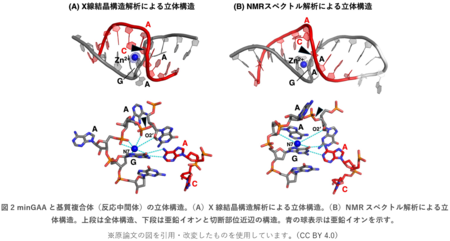
触媒メカニズムを解明するために、X線結晶構造解析およびNMRスペクトル解析による反応中間体の立体構造決定を行いました(図2)。前者は、亜鉛イオンの結合位置が明確にわかる一方で、結晶中の環境に構造が依存する傾向が高くなります(図2A)。逆に、後者は亜鉛イオンを直接には観察できませんが、溶液中の自然な立体構造を明らかにできます(図2B)。これらの補完的な手法を用いることでminGAAの立体構造は、いずれも全体的にB型構造と呼ばれる特徴を持ち、さらに触媒部のA塩基と基質のG塩基が塩基対を形成すること、亜鉛イオンはそのG塩基のN7原子と配位結合することが明らかになりました。また、水溶液中で亜鉛イオンには最大6個の水分子が配位結合しますが、これらの水分子と切断部位のO2′原子が接触できる位置関係でした。
【画像:https://kyodonewsprwire.jp/img/202601192637-O3-r009SMbj】
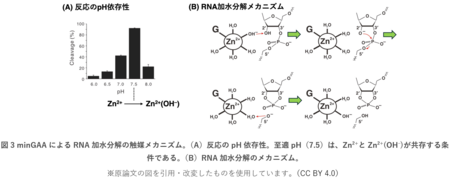
DNA酵素minGAAは、pH7.5で最大の活性を示します(図3)。この条件では、亜鉛イオンはZn2+(配位子は全てH2O)とZn2+(OH–)(配位子のうち一つがOH–)が同等の割合で共存します。触媒メカニズムにおいては、基質のG塩基と配位結合している亜鉛イオンが、Zn2+(OH–)となっている場合に、そのOH–から切断部位のO2′原子に負電荷が移動することで触媒反応が引き起こされると考えられます。このとき、RNAは切断されて末端は2′, 3′-環状リン酸の形となることが、質量分析によってわかります。負電荷はO5′原子を介して、再び亜鉛イオンの配位子に戻ります。
【画像:https://kyodonewsprwire.jp/img/202601192637-O4-thy6gHi4】
本研究で開発されたDNA酵素minGAAは、触媒部がACの2塩基のみという、これまでにない最小のDNA酵素です。一方、基質側の対応する部分もAAG(5′側から)の3塩基のみとなります。つまり、AAG配列を有するRNAであれば、その前後に合わせた相補的な配列をDNA酵素の基質認識部としてデザインすることで、意図する位置での切断が期待できます。長鎖mRNAを簡便かつ効率的に解析できる新たなツールとして、品質管理などへの応用が期待できます。
今後の予定
今後もインビトロ核酸選別法を利用してさまざまな機能性核酸分子を創出するとともに、分子メカニズム解明のための立体構造解析研究を展開する予定です。
論文情報
掲載誌: Nucleic Acids Research
論文タイトル:A minimal RNA-cleaving DNAzyme and its catalytic mechanism
著者:山崎和彦#*、猪股梨華#、山崎智子、久保田智巳、宮下尚之、竹内恒、宮岸真*
(#はequal contribution)
*責任著者:
国立研究開発法人産業技術総合研究所 健康医工学研究部門 キャリアリサーチャー、筑波大学グローバル教育院 教授 宮岸 真、
モレキュラーバイオシステム研究部門、主任研究員 山崎 和彦
DOI:10.1093/nar/gkaf1502
用語解説
DNA酵素(DNAzyme)
酵素活性を有するDNA分子であり、天然には存在しない。核酸を基質として切断あるいは融合するDNA酵素の場合、一般に、基質配列と(Aに対してT、Gに対してCなど)相補的な配列によって基質認識を行う基質認識部と、触媒作用を発揮する非相補性の触媒部からなる。前者は、基質の配列に合わせて任意のデザインが可能である。
インビトロ核酸選別法
A、C、G、T塩基(DNAの場合)のいずれかから構築される塩基配列をランダム化して、あらゆる組み合わせを包含するライブラリーからターゲット分子を選別する方法。例えば標的分子との結合をもとに選別するアプタマーなどが取得できる。分子進化法、SELEXとも呼ばれる。
NMRスペクトル解析
強磁場下では、1Hや13Cなどの原子核が特定の共鳴周波数に対応する電磁波(エネルギー)を吸収する。原子核を取り巻く電子環境によってこの共鳴周波数が変化するため、スペクトル上でピークとして分離して観測される。エネルギー吸収後の放出過程では、距離の近い原子核の間でエネルギー移動(核オーバーハウザー効果)が生じるため、こうした距離情報を多数集めることで、立体構造が決定される。
X線結晶構造解析
対象分子の結晶にX線を照射すると、分子中の電子との相互作用よって回折パターンが得られる。これらの回折強度を解析することで、電子密度分布が再構築される。得られた電子密度に分子モデルを当てはめることで、立体構造が決定される。
mRNA医薬
特定のタンパク質をコードする人工合成mRNAを細胞内に導入し、体内で当該タンパク質を発現させることで、治療・予防効果を得ることを目的としたワクチン等の医薬品。新型コロナウイルスのワクチンなど、数千ヌクレオチドのものを含む。
配列解析
核酸のヌクレオチド配列を決定する方法。DNA合成反応中に伸長停止を行って断片の長さを解析するサンガー法や多数の分子を一度に解析する次世代シークエンシング法によって行われる。
核酸アプタマー
標的とする分子に対して、抗体のように特異的に結合する人工的な核酸(DNAあるいはRNA)およびその修飾体。インビトロ核酸選別法により取得する。
B型構造
水溶液中のDNA二重鎖がとる代表的な構造で、塩基対がほぼ平行な梯子状に規則的に積み重なった右巻きのらせん構造を特徴とする。
配位結合
水や窒素化合物などの分子(配位子)が金属イオンに電子対を供与して生じる化学結合である。亜鉛イオンでは、通常4個(正四面体)から6個(正八面体)の配位子が結合する。
配位子
金属イオンなど中心原子に対して、配位結合により結合している水などの分子。
質量分析
分子やその断片をイオン化し、電場中での移動速度から質量電荷比(m/z)を測定する方法である。この情報は、化学構造を解析する上で重要である。
プレスリリースURL
https://www.aist.go.jp/aist_j/press_release/pr2026/pr20260121_2/pr20260121_2.html
プレスリリースURL
https://kyodonewsprwire.jp/release/202601192637
本プレスリリースは発表元が入力した原稿をそのまま掲載しております。詳細は上記URLを参照下さい。また、プレスリリースへのお問い合わせは発表元に直接お願いいたします。
